réservé à la recherche
JIB-04 Histone Demethylase inhibiteur
N° Cat.S7281
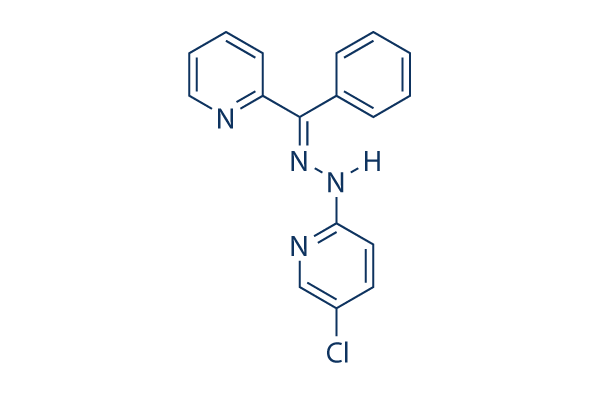
Structure chimique
Poids moléculaire: 308.76
Contrôle qualité
| Cibles apparentées | HDAC JAK BET Histone Methyltransferase PKC PARP HIF PRMT EZH2 AMPK |
|---|---|
| Autre Histone Demethylase Inhibiteurs | GSK-J4 Hydrochloride SP2509 GSK-LSD1 2HCl Ladademstat (ORY-1001) Dihydrochloride CPI-455 HCl OG-L002 IOX1 GSK J1 ML324 CPI-455 |
Informations chimiques, stockage et stabilité
| Poids moléculaire | 308.76 | Formule | C17H13ClN4 |
Stockage (À partir de la date de réception) | |
|---|---|---|---|---|---|
| N° CAS | 199596-05-9 | Télécharger le SDF | Stockage des solutions mères |
|
|
| Synonymes | NSC 693627 | Smiles | C1=CC=C(C=C1)C(=NNC2=NC=C(C=C2)Cl)C3=CC=CC=N3 | ||
Solubilité
|
In vitro |
DMSO
: 62 mg/mL
(200.8 mM)
Water : Insoluble Ethanol : Insoluble |
Calculateur de molarité
|
In vivo |
|||||
Calculateur de formulation in vivo (Solution claire)
Étape 1 : Entrez les informations ci-dessous (Recommandé : Un animal supplémentaire pour tenir compte des pertes pendant lexpérience)
Étape 2 : Entrez la formulation in vivo (Ceci nest que le calculateur, pas la formulation. Veuillez nous contacter dabord sil ny a pas de formulation in vivo dans la section Solubilité.)
Résultats du calcul :
Concentration de travail : mg/ml;
Méthode de préparation du liquide maître DMSO : mg médicament prédissous dans μL DMSO ( Concentration du liquide maître mg/mL, Veuillez nous contacter dabord si la concentration dépasse la solubilité du DMSO du lot de médicament. )
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuiteμL PEG300, mélanger et clarifier, ajouter ensuiteμL Tween 80, mélanger et clarifier, ajouter ensuite μL ddH2O, mélanger et clarifier.
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuite μL Huile de maïs, mélanger et clarifier.
Remarque : 1. Assurez-vous que le liquide est clair avant dajouter le solvant suivant.
2. Assurez-vous dajouter le(s) solvant(s) dans lordre. Vous devez vous assurer que la solution obtenue lors de lajout précédent est une solution claire avant de procéder à lajout du solvant suivant. Des méthodes physiques telles que le vortex, les ultrasons ou le bain-marie peuvent être utilisées pour faciliter la dissolution.
Mécanisme daction
| Targets/IC50/Ki |
JARID1A
(Cell-free assay) 230 nM
JMJD2D
(Cell-free assay) 290 nM
JMJD2E
(Cell-free assay) 340 nM
JMJD2B
(Cell-free assay) 435 nM
JMJD2A
(Cell-free assay) 445 nM
JMJD3
(Cell-free assay) 855 nM
|
|---|---|
| In vitro |
JIB-04 induit des changements transcriptionnels de manière sélective pour le cancer, y compris la régulation négative des gènes prolifératifs et la régulation positive des gènes anti-prolifératifs/pro-apoptotiques. Ce composé bloque la croissance des lignées de cancer du poumon et de la prostate avec une IC50 aussi faible que 10 nM, tout en produisant moins d'activités anti-prolifératives sur les HBEC et les PrSC/PrEC.
|
| Kinase Assay |
Tests d'activité de la Jumonji demethylase/prolyl hydroxylase/LSD1
|
|
La JMJD2E aa 1-350 active est purifiée à partir d'E.coli et utilisée in vitro en présence d'α-cétoglutarate, de 5-10 μM de fer, d'acide ascorbique et d'un substrat peptidique d'histone dans une réaction couplée avec la formaldéhyde déshydrogénase supplémentée en NAD+ pour quantifier la production de NADH ou en utilisant le kit Epigentek P-3081. Pour les réactions de déméthylation des histones quantifiées par analyse Western, un construit d'expression His-tagué hJMJ2D aa 1-350, aimablement fourni par les Drs. Y. Shi et J. Whetstine, est exprimé et purifié à partir d'E.coli selon les instructions du manuel Qiagen Ni-NTA agarose et le protocole de Whestine et al 54. En bref, la protéine est éluée dans 50 mM TrisHCl pH 7.8 + 0.3 M NaCl + 10% Glycérol + 200 mM immidazole, et dialysée contre 20 mM TrisHCl pH 7.4, 0.15 M NaCl, 0.2 mM PMSF, 0.5 mM DTT, 8% glycérol. L'enzyme est aliquoteé, congelée rapidement et stockée à -80°C. Pour les tests d'activité par Western blot, ~1.5 μg d'enzyme sont combinés avec 0.3 μg de substrat H3K9me3 (Active Motif #31213) dans 10 μM (NH4)2Fe(SO4)2, 1 mM d'α-cétoglutarate et 2 mM de L-ascorbate de sodium dans 50 mM Hepes pH 7.9 en présence de véhicule ou de médicament et incubés pendant 30 min-2 heures à 37°C. Le tampon de charge SDS est ajouté aux réactions, et après ébullition, les échantillons sont passés sur des gels NuPage 4-12% Bis-Tris, transférés sur nitrocellulose et blottés en utilisant Upstate #07-523 pour détecter H3K9me3. Pour la détection du signal total H3, nous utilisons Active Motif #39763 (primaire) et un anticorps secondaire de chèvre anti-lapin conjugué à l'IRDye 680 (secondaire, LI-COR # 926-32223) et les blots imagés dans un système d'imagerie infrarouge Odyssey aimablement mis à disposition par le Dr M. Cobb. Pour les déterminations in vitro d'IC50 et les études de compétition, typiquement 100-200 ng de protéine purifiée sont incubés avec le véhicule, JIB-04 ou des analogues, comme indiqué dans les légendes des figures et l'activité mesurée par ELISA (kit Epigentek P-3081 pour la déméthylation H3K9me3, P-3083 pour la déméthylation H3K4me3, et P-3085 pour la déméthylation H3K27me3) dans des réactions contenant 50mM Hepes pH 7.5, 0.01% Tween 20, 120nM (NH4)2Fe(SO4)2, 1 mM d'α-cétoglutarate, 2 mM de L-ascorbate de sodium et 50ng de substrat peptidique. Les concentrations finales d'enzymes dans les réactions étaient les suivantes : 206 nM JMJD2A, 12 nM JMJD2B, 60 nM JMJD2C, 90 nM JMJD2D E.coli, 30 nM JMJD2D Sf9, 30 nM JMJD2E, 30 nM Jarid1a, 35 nM JMJD3. Les lectures de fond sont données par des enzymes inactivées par la chaleur, 0.5-1 mM 2,4 PDCA ou des réactions sans 2-OG. hJMJD2A (aa1-350) purifié dans E.coli est le don aimable du Dr Jose Rizo-Rey et est dosé à 400 ng/réaction en raison de son activité intrinsèque faible. Le logiciel GraphPad Prism est utilisé pour les calculs d'IC50 et l'ajustement de courbes. E.coli JMJD2D purifiée par nous et Sf9 JMJD2D de BPS ont donné des résultats indiscernables. Notez que pour les tests de compétition de substrat, afin de rester dans la plage linéaire du test et de ne pas saturer la capacité de liaison de la plaque ELISA, les réactions avec > 0.75μM H3K9me3 contiennent un substrat non biotinylé ou sont diluées à l'étape de détection et les signaux ajustés par facteur de dilution. Pour la quantification directe de l'activité H3K9me3 déméthylase dans les lysats cellulaires, les cellules traitées (plaquées à 2 millions/boîte de 10 cm) ou les homogénats tumoraux dans du PBS ont été sonicés (3x 4 sec) et des quantités égales de protéine sont incubées avec un substrat d'histone H3K9me3 dans un tampon de réaction contenant des cofacteurs pendant 2h à 37°C avant une immunodétection spécifique du produit H3K9me2 en utilisant les réactifs du kit Epigentek P-3081. 500 ng de protéine PHD2 purifiée d'E.coli dans 40mM Tris pH 7.4, 100mM NaCl, 20% glycérol, 5mM β-mercapto-éthanol, 10mM maltose sont utilisés pour obtenir une activité dans la plage linéaire. Des peptides biotinylés dérivés de l'ODD de HIF-1 (Biotin-Acp-DLDLEALAPYIPADDDFQL ou Biotin-Acp-DLDLEALAP(OH)YIPADDDFQL comme contrôle hydroxylé) sont immobilisés sur des plaques de 96 puits revêtues de Neutravidine. L'enzyme est incubée dans les puits revêtus dans un tampon de réaction (20 mM Tris-Cl pH 7.5, 5 mM KCl, 1.5 mM MgCl2, 2 mM DTT, 0.12 μM de sulfate ferreux, 0.5 mM de 2-oxoglutarate et 1 mM d'ascorbate) pendant 45 min à température ambiante en présence des médicaments indiqués. L'analogue compétitif de l'α-cétoglutarate, le DMOG, est utilisé comme contrôle positif d'inhibition. L'hydroxylation des peptides est détectée en utilisant un anticorps polyclonal de lapin dirigé contre un épitope peptidique HIF hydroxylé (lapin anti-hydroxyproline 4817, fabriqué en interne), suivi de l'ajout d'un anticorps secondaire de chèvre anti-lapin conjugué à la HRP. La luminescence est mesurée dans un lecteur de plaques EnVision. L'activité de la protéine recombinante LSD1 est mesurée en utilisant le kit Epigentek P-3075 selon le protocole du fabricant avec l'inhibiteur propriétaire.
|
|
| In vivo |
Dans deux modèles de souris xénogreffes distincts (H358 ou A549), JIB-04 diminue la croissance tumorale, réduit l'activité de la Jumonji histone demethylase dans les tumeurs et prolonge la survie au cancer.
|
Références |
Applications
| Méthodes | Biomarqueurs | Images | PMID |
|---|---|---|---|
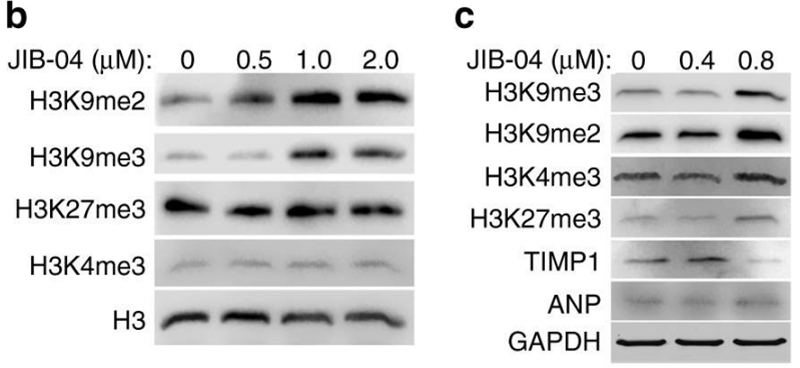
| Western blot | H3K9me2 / H3K9me3 / H3K27me3 / H3K4me3 / TIMP1 / ANP αCD133 |
|
30531796 |
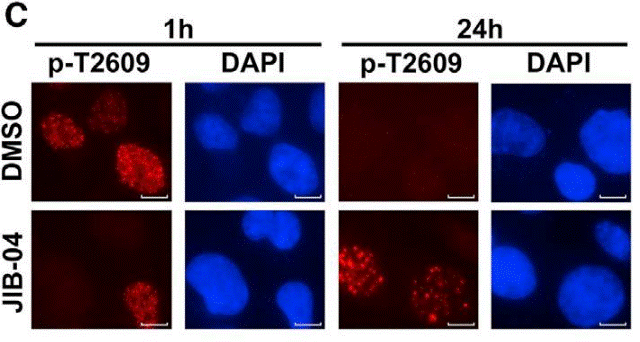
| Immunofluorescence | DNA-PKcs |
|
30355483 |
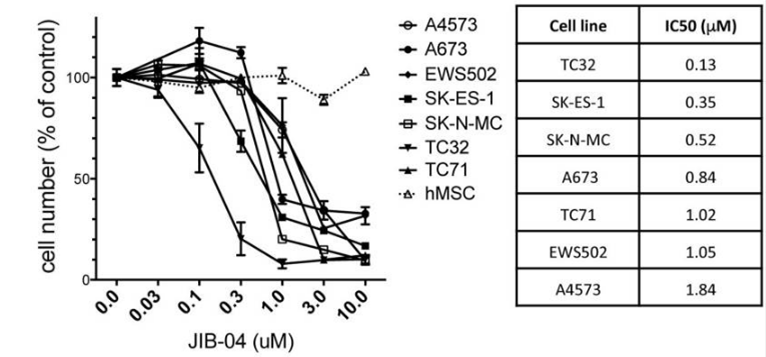
| Growth inhibition assay | Cell viability |
|
30237855 |
Support technique
Tel: +1-832-582-8158 Ext:3
Si vous avez dautres questions, veuillez laisser un message.
Questions fréquemment posées
Question 1:
Do you have any suggestion for in vivo study please? (via injection)
Réponse :
For IP or sub cutaneous injection, this compound is soluble in 4% DMSO+30% PEG 300+5% Tween 80+ddH2O at 3 mg/ml. But when water added, it turned yellowish green immediately. We are not sure if it was still fine in this vehicle. So we tried to use oil. 3mg of it dissolves in 40ul of DMSO clearly, and it can be dilute with corn oil at any proportion.
