réservé à la recherche
Crenolanib (CP-868596) Inhibiteur de PDGFR/FLT3
N° Cat.S2730
Structure chimique
Poids moléculaire: 443.54
Contrôle qualité
| Cibles apparentées | EGFR VEGFR JAK PDGFR FGFR Src HIF FLT HER2 Bcr-Abl |
|---|---|
| Autre FLT3 Inhibiteurs | UNC2025 Dovitinib (TKI-258) Dovitinib (TKI258) Lactate monohydrate Tandutinib (MLN518) KW-2449 ENMD-2076 AST-487 (NVP-AST487) TCS 359 G-749 AMG 925 |
Culture cellulaire, traitement et concentration de travail
| Lignées cellulaires | Type dessai | Concentration | Temps dincubation | Formulation | Description de lactivité | PMID |
|---|---|---|---|---|---|---|
| A549 cells | Cell viability assay | 3.9-1000 nM | 72 h | a dose-dependent inhibition of cancer cell viability | 25328409 | |
| MV4-11 | Function assay | a decrease in mitochondrial matrix protein aconitase hydratase 2 (ACO2) | 25328409 | |||
| MV4-11 | Cytotoxicity assay | Cytotoxicity against human MV4-11 cells expressing FLT3 ITD mutant, IC50=0.0015μM | 31207462 | |||
| MOLM13 | Cytotoxicity assay | Cytotoxicity against human MOLM13 cells expressing FLT3 ITD mutant, IC50=0.0049μM | 31207462 | |||
| MV4-11 | Cytotoxicity assay | 72 hrs | Cytotoxicity in human MV4-11 cells assessed as inhibition of cellular viability incubated for 72 hrs by CellTiter-Blue assay, IC50=0.012μM | 30742435 | ||
| DAOY | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for DAOY cells | 29435139 | |||
| SJ-GBM2 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for SJ-GBM2 cells | 29435139 | |||
| SK-N-MC | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for SK-N-MC cells | 29435139 | |||
| BT-37 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for BT-37 cells | 29435139 | |||
| NB-EBc1 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for NB-EBc1 cells | 29435139 | |||
| U-2 OS | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for U-2 OS cells | 29435139 | |||
| Saos-2 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for Saos-2 cells | 29435139 | |||
| LAN-5 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for LAN-5 cells | 29435139 | |||
| BT-12 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for BT-12 cells | 29435139 | |||
| Rh18 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for Rh18 cells | 29435139 | |||
| OHS-50 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for OHS-50 cells | 29435139 | |||
| RD | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for RD cells | 29435139 | |||
| A673 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Confirmatory screen for A673 cells) | 29435139 | |||
| DAOY | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Confirmatory screen for DAOY cells | 29435139 | |||
| U-2 OS | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Confirmatory screen for U-2 OS cells | 29435139 | |||
| Rh41 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Confirmatory screen for Rh41 cells | 29435139 | |||
| SK-N-SH | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Confirmatory screen for SK-N-SH cells | 29435139 | |||
| MV4-11 | Cytotoxicity assay | 0.5 to 5000 nM | 2 to 8 hrs | Cytotoxicity in human MV4-11 cells assessed as inhibition of cellular viability at 0.5 to 5000 nM incubated for 2 to 8 hrs followed by compound washout by CellTiter-Glo luminescent assay | 30742435 | |
| Cliquez pour voir plus de données expérimentales sur les lignées cellulaires | ||||||
Informations chimiques, stockage et stabilité
| Poids moléculaire | 443.54 | Formule | C26H29N5O2 |
Stockage (À partir de la date de réception) | |
|---|---|---|---|---|---|
| N° CAS | 670220-88-9 | Télécharger le SDF | Stockage des solutions mères |
|
|
| Synonymes | CP-868596, ARO 002 | Smiles | CC1(COC1)COC2=CC3=C(C=C2)N(C=N3)C4=NC5=C(C=CC=C5N6CCC(CC6)N)C=C4 | ||
Solubilité
|
In vitro |
DMSO
: 88 mg/mL
(198.4 mM)
Ethanol : 88 mg/mL Water : Insoluble |
Calculateur de molarité
|
In vivo |
|||||
Calculateur de formulation in vivo (Solution claire)
Étape 1 : Entrez les informations ci-dessous (Recommandé : Un animal supplémentaire pour tenir compte des pertes pendant lexpérience)
Étape 2 : Entrez la formulation in vivo (Ceci nest que le calculateur, pas la formulation. Veuillez nous contacter dabord sil ny a pas de formulation in vivo dans la section Solubilité.)
Résultats du calcul :
Concentration de travail : mg/ml;
Méthode de préparation du liquide maître DMSO : mg médicament prédissous dans μL DMSO ( Concentration du liquide maître mg/mL, Veuillez nous contacter dabord si la concentration dépasse la solubilité du DMSO du lot de médicament. )
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuiteμL PEG300, mélanger et clarifier, ajouter ensuiteμL Tween 80, mélanger et clarifier, ajouter ensuite μL ddH2O, mélanger et clarifier.
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuite μL Huile de maïs, mélanger et clarifier.
Remarque : 1. Assurez-vous que le liquide est clair avant dajouter le solvant suivant.
2. Assurez-vous dajouter le(s) solvant(s) dans lordre. Vous devez vous assurer que la solution obtenue lors de lajout précédent est une solution claire avant de procéder à lajout du solvant suivant. Des méthodes physiques telles que le vortex, les ultrasons ou le bain-marie peuvent être utilisées pour faciliter la dissolution.
Mécanisme daction
| Targets/IC50/Ki |
FLT3
(CHO cells) 0.74 nM(Kd)
PDGFRα
(CHO cells) 2.1 nM(Kd)
PDGFRβ
(CHO cells) 3.2 nM(Kd)
|
|---|---|
| In vitro |
Le Crenolanib est significativement plus puissant pour inhiber l'activité kinase des kinases PDGFRα résistantes (D842I, D842V, D842Y, D1842-843IM et délétion I843). Ce composé est plus puissant contre D842V dans le système modèle isogénique, avec une IC50 d'environ 10 nM. Il inhibe l'activité kinase de l'oncogène de fusion dans la lignée cellulaire EOL-1, qui est dérivée d'un patient atteint de leucémie éosinophile chronique et exprime la kinase de fusion FIP1L1-PDGFRα constitutivement activée, avec une IC50 = 21 nM. Cette substance chimique inhibe également la prolifération des cellules EOL-1 avec une IC50 = 0,2 pM. Elle inhibe l'activation des kinases mutantes V561D ou D842V exprimées dans les cellules BaF3 avec une IC50 de 85 nM ou 272 nM, respectivement. Ce composé inhibe l'activation de PDGFRα dans la lignée cellulaire de cancer du poumon non à petites cellules H1703 qui présente une amplification de 24 fois de la région 4q12 contenant le locus PDGFRα, avec une IC50 de 26 nM. C'est un inhibiteur de TKI PDGFR hautement puissant et sélectif, biodisponible par voie orale. Ce composé de benzimidazole a des IC50 de 0,9 nM et 1,8 nM pour PDGFRA et PDGFRB, respectivement. |
| Kinase Assay |
Évaluation biochimique de l'activité kinase de PDGFRα
|
|
Des cellules ovariennes de hamster chinois (CHO) sont transfectées transitoirement avec des constructions PDGFRα mutées ou de type sauvage et traitées avec diverses concentrations de Crenolanib. Les expériences impliquant de l'ADN recombinant sont réalisées dans des conditions de niveau de biosécurité 2 conformément aux directives. Des lysats protéiques de lignées cellulaires sont préparés et soumis à une immunoprécipitation à l'aide d'anticorps anti-PDGFRα, suivie d'un immunobuvardage séquentiel pour PDGFRα. La densitométrie est réalisée pour quantifier l'effet du médicament à l'aide du logiciel Photoshop, le niveau de phosphor-PDGFRα étant normalisé par rapport à la protéine totale. Les résultats expérimentaux de densitométrie et de prolifération sont analysés à l'aide du logiciel Calcusyn 2.1 pour déterminer mathématiquement les valeurs d'IC50. Le test de somme des rangs de Wilcoxon est utilisé pour comparer les valeurs d'IC50 de ce composé pour une mutation donnée.
|
|
| In vivo |
Ce composé, un inhibiteur de PDGFR, supprime la prolifération des cellules cancéreuses du poumon et inhibe la croissance tumorale in vivo |
Références |
|
Applications
| Méthodes | Biomarqueurs | Images | PMID |
|---|---|---|---|
| Western blot | pKIT / KIT pFLT3 / FLT3 / pERK / ERK / pS6 / S6 pAKT / AKT / pMAPK / MAPK / pSTAT5 / STAT5 |

|
24623852 |
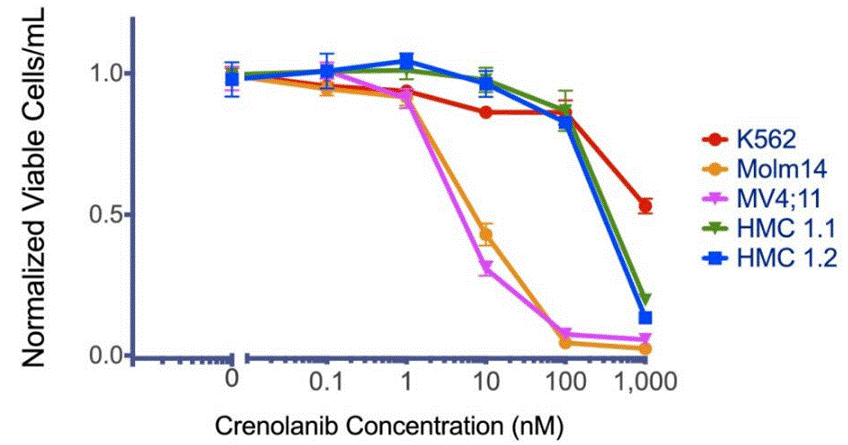
| Growth inhibition assay | Cell viability |
|
24623852 |
Informations sur lessai clinique
(données de https://clinicaltrials.gov, mis à jour le 2024-05-22)
| Numéro NCT | Recrutement | Conditions | Sponsor/Collaborateurs | Date de début | Phases |
|---|---|---|---|---|---|
| NCT00949624 | Completed | Advanced Solid Tumors |
Arog Pharmaceuticals Inc. |
December 2005 | Phase 1 |
Support technique
Tel: +1-832-582-8158 Ext:3
Si vous avez dautres questions, veuillez laisser un message.
